Dra. Irene Luque Fernández
- Categoría Profesional Actual: Catedrático de Universidad.
- Código ORCID: 0000-0003-2757-4779
- Correo Electrónico de contacto: iluque@ugr.es
- Teléfono de contacto: +34958240440
- Directorio UGR: Acceso Información institucional directorio UGR
Breve Resumen de la Carrera Investigadora de la Dra. Luque Fernández:
Irene Luque Fernández es catedrática, directora del Departamento de Química Física y miembro del Instituto de Biotecnología y de la Unidad de Excelencia de Química Aplicada a Biomedicina y Medioambiente de la Universidad de Granada (UGR). Obtuvo el doctorado en Química por la UGR en 1998 sobre el desarrollo de algoritmos para el diseño racional de ligandos peptídicos y su aplicación a la inhibición de la proteasa del VIH-1 y la elucidación de origen molecular de la resistencia a fármacos. Los algoritmos desarrollados se protegieron bajo patente, licenciada a varias compañías farmacéuticas. Entre 1998 y 2001 realizó una estancia postdoctoral en el laboratorio del Dr. Ernesto Freire (Johns Hopkins University, EEUU), abordando el estudio termodinámico-estructural de las bases moleculares de la cooperatividad, alosterismo y transducción de señales en proteínas y de la inhibición de las proteasas aspárticas del VIH y Plasmodium Falciparum, como dianas frente al SIDA y la Malaria. En 2002 se incorporó al Departamento de Química Física de la UGR como investigadora Ramón y Cajal. Desde entonces su trabajo ha estado enfocado fundamentalmente en el estudio termodinámico y estructural del reconocimiento de secuencia ricas en prolina por parte de dominios modulares de interacción proteína-proteína, combinando aproximaciones experimentales y computacionales, centrándose en los dominios SH3 de los oncogenes Src, Yes y Abl, en los dominios WW del oncogén YAP65 y en las dianas celulares de los dominios L víricos (Tsg101-UEV, Nedd4-WW3 y Alix-V), con el objetivo de identificar nuevos agentes antitumorales y antivirales. Su investigación ha permitido avanzar en la comprensión de la naturaleza del reconocimiento de secuencias ricas en prolina, estableciendo un nuevo paradigma de unión en el que las interacciones mediadas por moléculas de agua y la elevada plasticidad conformacional de los dominios juegan un papel determinante en la afinidad y especificidad. Su equipo ha demostrado que, a pesar de la promiscuidad general de estas interacciones, es posible identificar ligandos con alta afinidad y buen perfil de especificidad. Esta línea de trabajo ha sido financiada con más 1.1 M de euros en cuatro proyectos del Plan Nacional de Investigación (convocatorias 2006, 2009, 2012 y 2016), de los que la Dra. Luque ha sido I. P. Ha sido, asimismo, responsable del paquete de trabajo de diseño en el proyecto EURONEUT41, financiado por la UE con 12 M de Euros y dedicado al desarrollo de una vacuna frente al SIDA basada en GP41, cuyos resultados han sido patentados. Actualmente su investigación persigue el desarrollo de inhibidores de la gemación vírica con buenas características farmacológicas, buen perfil de especificidad y baja toxicidad, como nuevos antivirales de amplio espectro. Para ello, como complemento a las metodologías biofísicas y computacionales clásicas, ha incorporado a su investigación técnicas de selección de péptidos por “phage display” y de cribado masivo de grandes colecciones de compuestos, lo que ha permitido identificar varias moléculas inhibidoras de la gemación vírica con potencial como nuevos antivirales de amplio espectro. Participa, asimismo, en un proyecto multidisciplinar dedicado a la búsqueda de inhibidores de Fascina como nuevos antimetastásicos, en el contexto del cual se han identificado algunas moléculas de interés, que han sido protegidas por una patente internacional, licenciada a ENNAID Therapeutics.
Líneas de Investigación desarrolladas por el equipo de la Dra. Luque Fernández
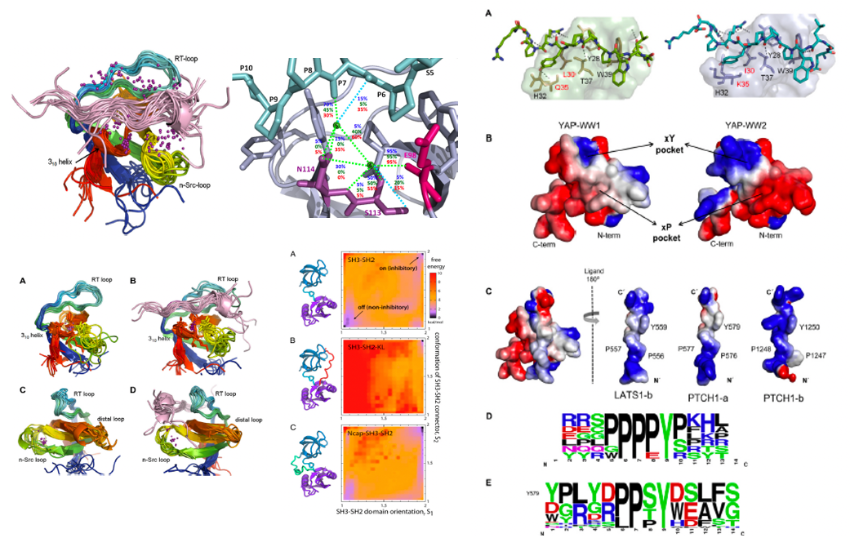
Estudio de la estabilidad, cooperatividad e interacción con ligandos de dominios modulares de reconocimiento de secuencias ricas en prolina.
Esta línea de investigción se centra en el estudio multidisciplinar del reconocimiento de secuencias ricas en prolina por dominios modulares de interacción proteína-proteína. En nuestro trabajo combinamos a) estudios biofísicos de la estabilidad, cooperatividad y afinidad de unión mediante técnicas espectroscópicas y calorimétricas; b) determinación estructural mediante difracción de rayos X y Resonancia Magnética Nuclear; c) estudios proteómicos mediante phage display y d) simulaciones de dinámica molecular, con el fin de elucidar las bases moleculares de la afindiad y especificidad de la interacción como base para el diseño de inhibidores de interés biomédico y biotecnológico. Nuestro trabajo se ha centrado en los dominios SH3 de los oncogenes Src, Yes y Abl, en los dominios WW del oncogén YAP65 y en las dianas celulares de los dominios L víricos Tsg101-UEV y Nedd4-WW3.
Esta línea de trabajo nos ha permitido avanzar en la comprensión de la naturaleza del reconocimiento de secuencias ricas en prolina, estableciendo un nuevo paradigma de unión en el que las interacciones mediadas por moléculas de agua y la elevada plasticidad conformacional de los dominios juegan un papel determinante en la afinidad y especificidad. Hemos podido demostrar que, a pesar de la promiscuidad general de estas interacciones, es posible identificar ligandos con alta afinidad y buen perfil de especificidad con potencial como base para el desarrollo de fármacos antivirales y antitumorales.
Identificación y desarrollo de antivirales de amplio espectro dirigidos al hospedador: reposición de fármacos y productos naturales.
Las enfermedades víricas son una de las principales causas de mortalidad a nivel mundial e implican enormes costes sociales y económicos. Para la mayoría de ellas se carece de vacunas o tratamientos eficaces, por lo que el desarrollo de nuevos antivirales es una prioridad. De especial urgencia es la adquisición de herramientas para la intervención temprana frente a epidemias causadas por patógenos emergentes especialmente virulentos y contagiosos. Este es el caso de los virus de ARN Ebola o Marburgo, causantes de fiebres hemorrágicas con una alta tasa de mortalidad. Las epidemias/pandemias que recurrentemente hemos sufrido en la última década causadas por virus zoonóticos (SARS/2002, MERS/2012, Ebola/2014, ZIKA/2016 and SARS-CoV-2/2019) han puesto en evidencia nuestra gran vulnerabilidad frente a la potencial aparición de nuevos virus que, en el contexto de un medio natural degradado y en una sociedad globalizada, tienen un gran potencial para convertirse rápidamente serias amenazas globales de efectos devastadores, como estamos actualmente experimentando con la actual pandemia por COVID19. Existe, por tanto, una necesidad urgente e inaplazable de desarrollar nuevos antivirales de amplio espectro que proporcionen herramientas eficaces de intervención temprana frente a patógenos emergentes.
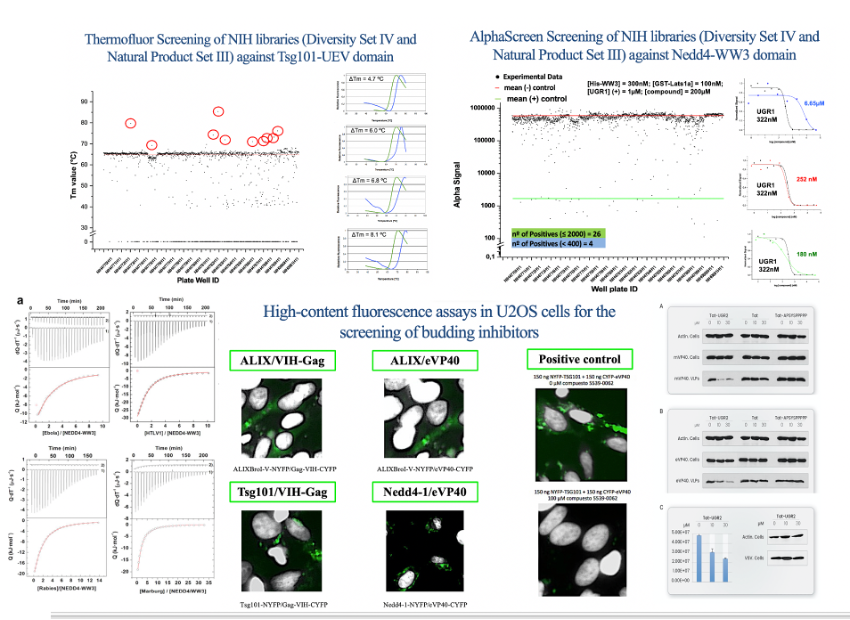
Para el secuestro de la maquinaria celular necesaria para su supervivencia, los virus establecen interacciones entre sus proteínas y proteínas humanas, que, en ocasiones se encuentra muy conservadas entre distintas familias de virus. La identificación de inhibidores que bloqueen estas interacciones es una estrategia prometedora para el desarrollo de nuevos antivirales de amplio espectro, activos frente a múltiples virus y con baja susceptibilidad al desarrollo de resistencia. En este contexto, estamos trabajando para el desarrollo de nuevos antivirales de amplio espectro dirigidos a proteínas del hospedador. Concretamente, nos centramos en dos líneas de acción:
- la búsqueda de moléculas que bloqueen las interacciones entre secuencias conservadas en múltiples virus denominadas Late domains y sus dianas celulares (Tsg101, NEDD4 y ALIX). Inhibidores de estas interacciones, que son esenciales para la gemación del virus de la célula infectada, han desmostrado su potencial antiviral frente a diferentes familias de virus, incluyendo retrovirus (VIH), filovirus (Ebola, Marburgo), rabdovirus (Rabia), etc.
- La identificación de inhibidores de las interacciónes proteína-proteína responsables de la respuesta inmune exacerbada asociada a la infección por SARS-CoV-2. Más específicamente de las interacciones entre la proteína Envelope (SARS-CoV-2-E) del virus y los dominios PDZ de la proteína humana sintenina, así como de la interacción entre la región N-terminal de sintenina y el dominio V de ALIX.
Para ello hemos puesto a punto una metodología multidisciplinar que combina el cribado virtual de grandes colecciones de compuestos, el cribado de alto rendimiento in vitro e in vivo (en ensayos celulares de alto contenido) y el estudio biofísico y estructural detallado de las dianas y sus interacciones con ligandos e inhibidores. Abordaremos el cribado no sólo de grandes colecciones de compuestos puros, sino también de colecciones de reposición de fármacos y de la colección de extractos microbianos de la Fundación MEDINA, una de las mayores y más diversas químicamente del mundo. Esto nos permitirá identificar potenciales inhibidores que serán testados utilizando ensayos in vitro, ensayos celulares así como estudios con virus vivos para validad su actividad antiviral.
Esta línea de investigación se desarrolla en colaboración con el área de “Cribado y validación de dianas” de la Fundación MEDINA, que dispone de instalaciones de primer nivel para el desarrollo de los cribados de alto rendimiento y con el grupo de investigación “Structural bioinformatics and high performance” liderado por el Dr. Horacio Pérez Sánchez en la Universidad Católica de Murcia.
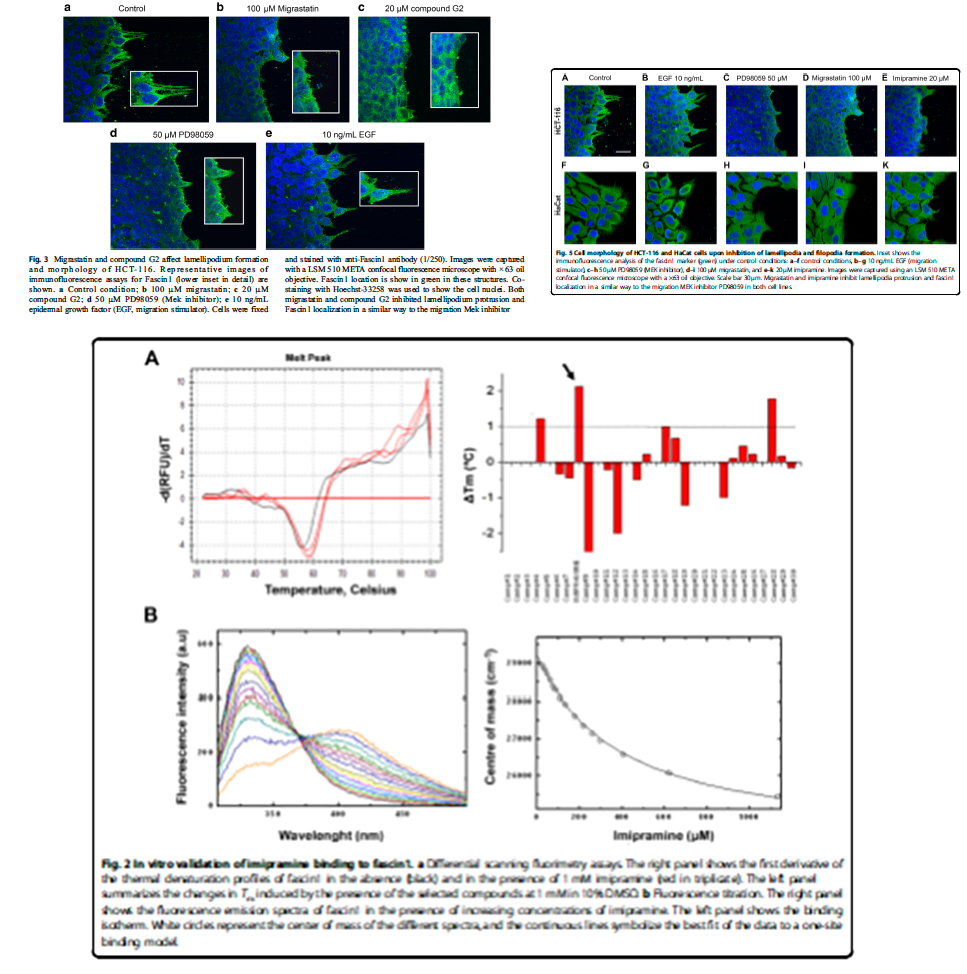
Inhibidores alostéricos de Fascina como nuevos fármacos antimetastásicos.
La diseminación por metástasis de las células tumorales es responsable del 90% de las muertes por cáncer. El desarrollo de nuevas terapias dirigidas específicamente a inhibir la migración celular y que complementen a los actuales tratamientos antiproliferativos es una necesidad médica prioritaria e ineludible. Fascina es una proteína expresada exclusivamente en células cancerígenas y encargada del empaquetamiento de los haces de Actina para la formación de filopodios e invadopodios en los procesos de migración e invasión celular. En la última década, Fascina se ha consolidado como un biomarcador de carcinomas agresivos y como una diana atractiva para el desarrollo de nuevos fármacos anti-metastásicos. Se ha demostrado que moléculas que bloquean la actividad de Fascina son potentes inhibidores de la migración, invasión y metástasis de células tumorales. Esto ha despertado un creciente interés por el desarrollo de nuevos inhibidores de Fascina con buenas propiedades farmacológicas. Estudios estructurales recientes han revelado que la introducción de mutaciones desactivantes o la unión de inhibidores inducen cambios en la estructura de Fascina, por lo que esta proteína es un buen candidato para la inhibición alostérica. Los fármacos alostéricos ofrecen grandes ventajas con respecto a fármacos convencionales dirigidos a sitios ortostéricos, ya que con frecuencia presentan mejor selectividad y propiedades farmacodinámicas, así como menores efectos secundarios. La mayoría de los fármacos alostéricos conocidos han sido identificados de forma fortuita en el contexto de cribados de alto rendimiento, por lo que el desarrollo de estrategias específicas para la búsqueda racional de moduladores alostéricos es de gran interés.
Durante los últimos cuatro años hemos participado en un proyecto multidisciplinar en colaboración con el grupo de investigación “Patología Molecular y Farmacogenética del Instituto Murciano de Investigación Biosanitaria, liderado por el Dr. Pablo Conesa Zamora, el grupo “Structural bioinformatics and high performance” liderado por el Dr. Horacio Pérez Sánchez en al Universidad Católica de Murcia, y el área de “Cribado y validación de dianas” de la Fundación MEDINA, lideradas por la Dra. Francisca Vicente Pérez. Esta colaboración se ha plasmado en la identificación de varios inhibidores de Fascina con actividad anti-migratoria y anti-invasiva, conjugando i) el cribado masivo in silico e in vitro de bibliotecas de compuestos ii) la caracterización biofísica de la diana y sus interacciones y iii) estudios funcionales en modelos celulares y animales.
En la actualidad, aprovechando las sinergias ya establecidas en este equipo multidisciplinar, nuestro objetivo es desarrollar una sólida base metodológica que permita racionalizar el descubrimiento de inhibidores alostéricos de Fascina con potencial como nuevos fármacos anti-metastásicos.
Para ello combinaremos un estudio detallado de las propiedades conformacionales de Fascina, combinando técnicas biofísicas (calorimetría diferencial de barrido y diversas técnicas espectroscópicas) con metodologías computacionales avanzadas (simulaciones de dinámica molecular o análisis de modos normales de vibración), que permita identificar las conformaciones y sitios alostéricos más relevantes para abordar la búsqueda de inhibidores alostéricos de Fascina mediante cribado virtual.
De forma complementaria, exploraremos la posibilidad de utilizar ensayos tipo Thermofluor (fluorescencia diferencial de barrido), ampliamente utilizados para la búsqueda de ligandos ortostéricos, para racionalizar la búsqueda de inhibidores alostéricos en campañas de cribado masivo de colecciones de compuestos puros y extractos microbianos de la colección de la Fundación MEDINA Además del potencial descubrimiento de nuevos inhibidores alostéricos de Fascina, esperamos generar una sólida base para racionalizar el proceso de búsqueda de nuevos fármacos seguros y eficaces frente a cánceres agresivos que no responden a los tratamientos tradicionales.